公募研究
 松井貴輝
松井貴輝
奈良先端科学技術大学院大学・バイオサイエンス研究科
〒630-0101奈良県生駒市高山町8916-5
TEL: 0743-72-5472
FAX: 0743-72-5479
E-mail: matsui@bs.naist.jp
研究室ホームページ:http://bsw3.naist.jp/courses/courses308.html
<略歴>
1996年:横浜市立大学 文理学部 卒業, 1998年:横浜市立大学大学院 総合理学研究科 修了, 2001年: 東京大学大学院 農学生命科学研究科 修了 博士(農学)取得, 2001年-2002年: 東京大学分子生物学研究所 宮島 篤 教授のもとでポスドク, 2002年-2005年: 米国ソーク研究所Juan Carlos
Izpisúa Belmonte教授のもとでリサーチアソシエイト, 2005年より、奈良先端科学技術大学院大学、バイオサイエンス研究科、助教
<研究課題>
A02 細胞集団形成過程における細胞と場のクロストーク
<研究計画>
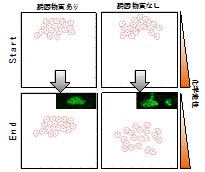
ゼブラフィッシュにおいて、クッペル胞(KV)は左右非対称性を規定するために重要な器官である。我々はこれまでに、FGFシグナルが細胞接着を増強することで、KV前駆細胞の集団形成を制御していることを発見した。最近我々は、接着にゆらぎがあること、接着がFGFシグナルを活性化する正のフィードバック効果を持つことを見出した(未発表)。これらの結果は、KV前駆細胞の集団形成は、「細胞」が取り囲む「場」を作り、その「場」が「細胞」シグナルを活性化するという「細胞」と「場」のクロストークによって制御されていることを示唆している。そこで本研究では、KV形成を器官形成のモデル系として、どのように正常な器官が形成されるのか、特に、細胞の動きに着目して解析する。具体的には、個々のKV前駆細胞と集団の動きを高解像度でイメージングし集団移動時に観察される変化パラメータを抽出し数理解析に用いる。また、細胞移動や接着レベルを改変したゼブラフィッシュ胚を用いることで、細胞移動と器官形成の関連性に迫りたい。
<イメージ図>
<代表的論文>
(1) Takaaki Matsui1, Akihiro Sasaki, Naoko Akazawa, Hifumi Otani and Yasumasa Bessho (2012) Celf1 regulation of dmrt2a is required for somite symmetry and left-right patterning during zebrafish development Development, 139: 3553-3560.
(2) Woong Kim, Takaaki Matsui, Masataka Yamao, Makoto Ishibashi, Kota Tamada, Toru Takumi, Kenji Kohno, Shigeyuki Oba, Shin Ishii, Yuichi Sakumura and Yasumasa Bessho (2011) The period of the somite segmentation clock is sensitive to Notch activity Molecular Biology of the Cell, 18: 3541-3549.
(3) Takaaki Matsui1, Siripong Thitamadee, Tomoko Murata, Hisaya Kakinuma, Takuji Nabetani, Yoshio Hirabayashi, Yoshikazu Hirate, Hitoshi Okamoto, and Yasumasa Bessho (2011) Canopy1, a positive feedback regulator of FGF signaling, controls progenitor cell clustering during Kupffer’s vesicle organogenesisProc. Natl. Acad. Sci. USA, 108: 9881-9886.
(4) Takaaki Matsui, Angel Raya, Yasuhiko Kawakami, Carles Callol-Massot, Javier Capdevila, Concepción Rodríguez Esteban, and Juan Carlos Izpisúa Belmonte (2005) Noncanonical Wnt signaling regulates midline convergence of organ primordia during zebrafish development Genes & Development, 19: 164-175
