
タンパク質の機能を理解する上で、その三次構造 (立体構造) から得られる情報は非常に多い。X線構造解析などにより明らかになった三次構造はデータベースに納められている。ここではその三次構造情報の入手法と得られた三次構造データを表示するプログラムの使用法について概説し、最後に立体構造予測についても簡単に述べる。
PDBのホームページにアクセス (http://pdb.protein.osaka-u.ac.jp/pdb/)
中央のサーチボックスにキーワードまたはPDB ID (各構造データに割り当てられている名前) を入力し、"Search" ボタンをクリック。

検索結果が表示される。適当なデータを選び、そのPDB IDをクリック。
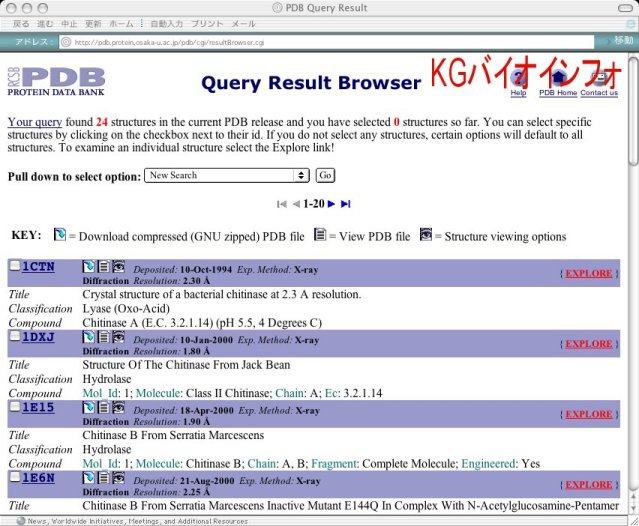
詳細な情報が表示される。データをダウンロードするには "Download/Display File" のボタンをクリック。
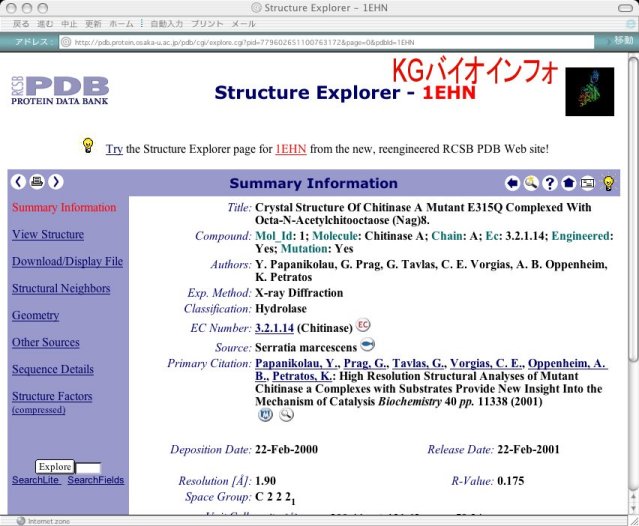
画面下部に非圧縮または圧縮されたPDBファイルをダウンロードするためのボタンがある。適当な解凍ソフトがある場合には圧縮されたファイルを、無い場合には非圧縮ファイルをダウンロードする。
上述の手順1でダウンロードしたデータはワードなどの文書ファイルとして開くこともできるが、専用の立体構造表示ソフトを使用する事により三次元的な表示が可能である。今回は "RasMol" というソフトを使用する。
RasMolソフトが無い場合、RasMolホームページからダウンロードする。 (http://umass.edu/microbio/rasmol/index2.htm)
RasMolソフトを立ち上げ、 "File" → "Open" で目的のPDBファイルを開く (PDBファイルの入っているフォルダ及び上位のフォルダ名に日本語が入っていると上手く認識されない場合がある)。
メインウィンドウに分子構造が表示される。画面内をドラッグすることで分子を回転させて異なる角度から見る事ができる。"Display", "Colours", "Options" などのプルダウンメニュー内の項目を変更することにより表示形式を様々に変える事が可能である。また、もう一つのウインドウ "Command Line" にコマンドを打ち込む事によって、特定のアミノ酸残基を指定して色や表示形式を変える事ができる。以下のホームページに丁寧な解説があるので参照されたい。
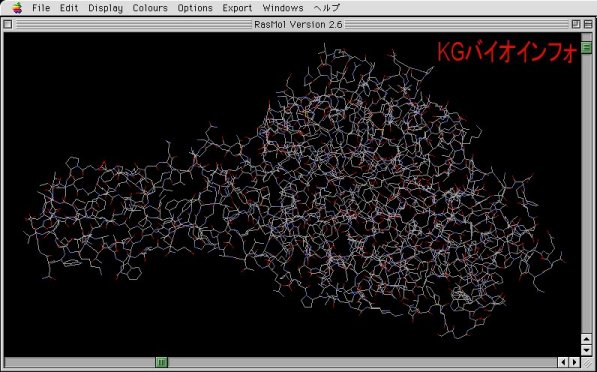
Display: Wireframe, Colours: CPK
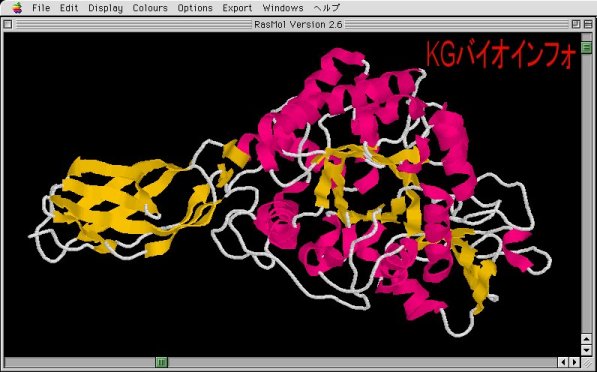
Display: Cartons, Colours: Structure
タンパク質の三次構造はその一次構造により規定されると考えられることから、一次構造から三次構造を予測する研究が精力的になされているが、現在のところ実用に耐えうるものは存在していない。だが、類縁のタンパク質で既に三次構造が判明しているものがあれば、それを鋳型に利用して三次構造を予測することは可能であり、ホモロジーモデリングと呼ばれている。これにより、三次構造が決定されていないタンパク質でも(すべてではないが)、立体構造からの考察を行うことが可能となってきている。しかし得られる情報はあくまで「予測」であり、現実とは異なる可能性があることに注意しなければならない。
SWISS-MODELのホームページ (http://swissmodel.expasy.org//SWISS-MODEL.html) にアクセスし、左メニュー画面の "First Approach mode" をクリック。
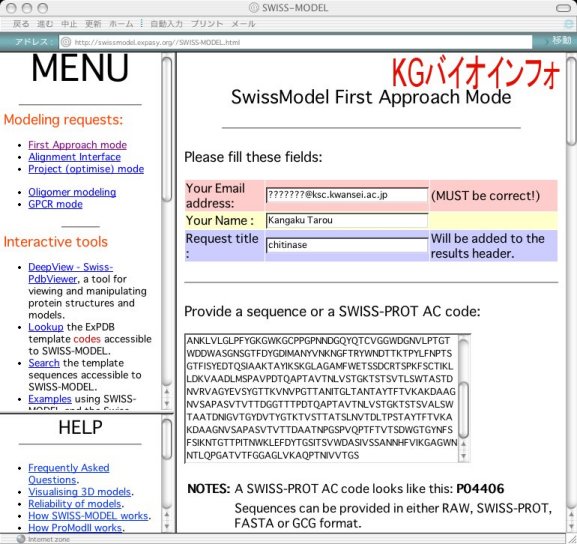
自分のメールアドレス、名前、配列名、アミノ酸配列をそれぞれ対応するボックスに記入する。
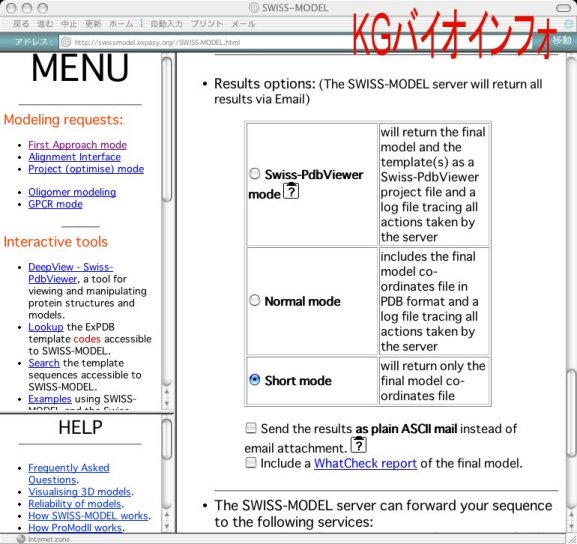
画面下部に進み、Results optionsの項目で "Short mode" を選択し、上画面の "Send Request" ボタンをクリックすると、5〜10分後に予測構造ファイルがメールに添付されて届く。
メールに添付された書類を "RasMol" などの立体構造表示ソフトで開く。送付配列の全体と相同性を示す配列がデータベースに存在しない場合には、分子の途中までしかモデリングされなかったり、二つに分割された結果が得られることがある。また、モデルのアミノ酸残基番号は送付したものに対応していない点も注意が必要である。