前出のblastxが (塩基配列) 対 (アミノ酸配列) のホモロジー検索であったのに対し、blastpは (アミノ酸配列) 対 (アミノ酸配列) のホモロジー検索である。NCBIのblastpではホモロジー検索を行うと同時にその蛋白質のドメイン構造に関する情報も得ることができ、大変便利である。ここでは大腸菌のDNAポリメラーゼIの配列を例に具体的な操作方法を説明する。
NCBIのBLASTのホームページ (http://www.ncbi.nlm.nih.gov/BLAST/) から"Protein-protein BLAST (blastp)"をクリックする。
Searchボックスにアミノ酸配列をペーストし、"BLAST!"ボタンをクリックして検索を開始する。
結果待ち受け画面が別のウインドウで開く。
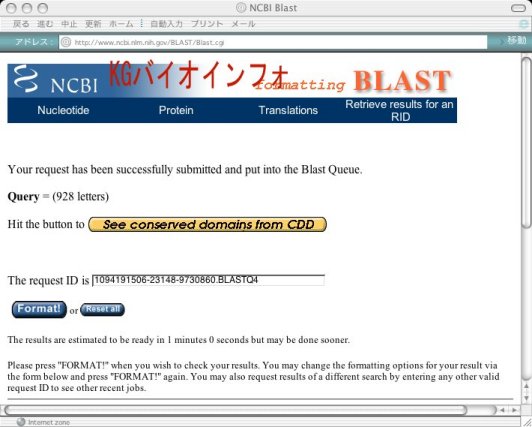
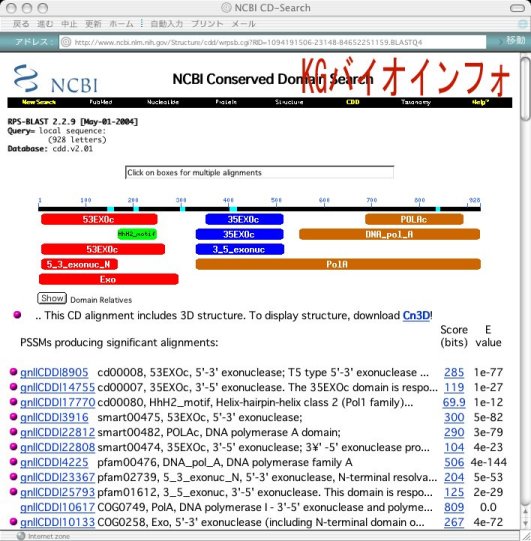
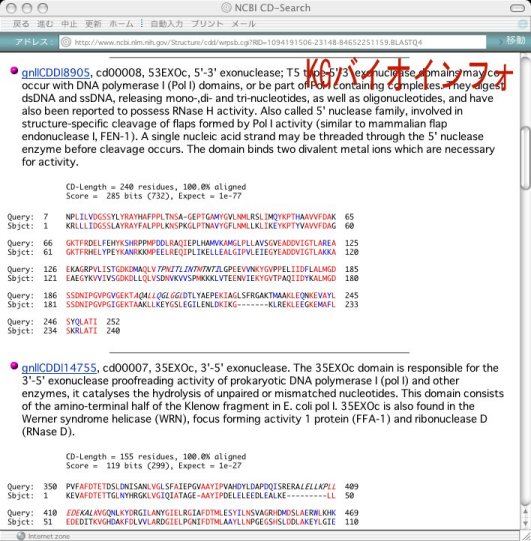
類似するドメインがドメインデータベースに存在するとその模式図もしくは上図のように"See conserved domain from CDD"のボタンが現れる。これをクリックすることにより、ドメインの詳細な情報を見ることができる (下図) 。
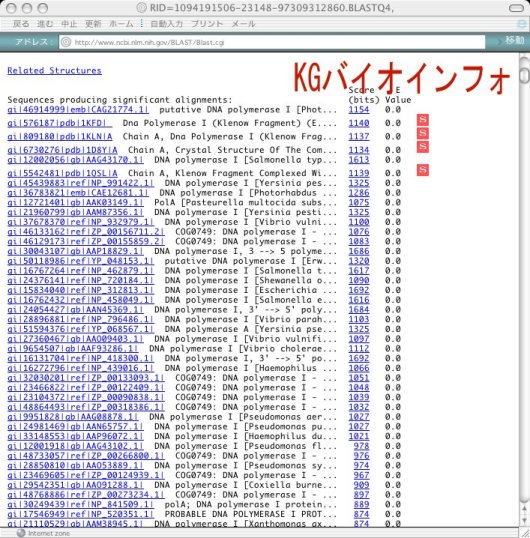
結果待ち受け画面の"Format!"ボタンをクリックすると別画面で結果が開く。問い合わせ配列中のどの部分が類似領域となっているかが画像で表される。図中ではすべて赤色となっているが、類似性の高い順に、赤-紫-緑-青-黒というように色分けされる。
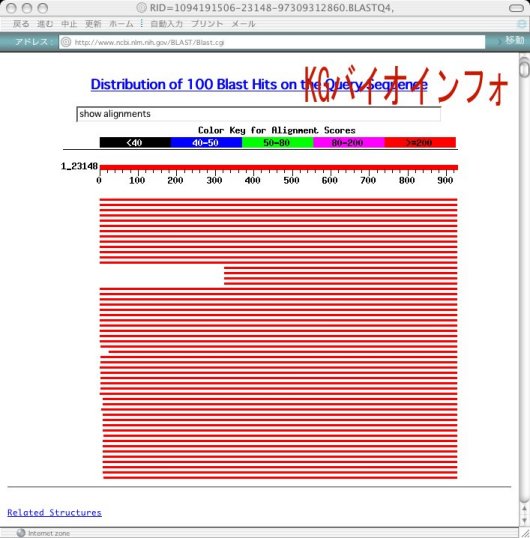
ウインドウ下部へ進むと類似性を示した配列の一覧が表示される。
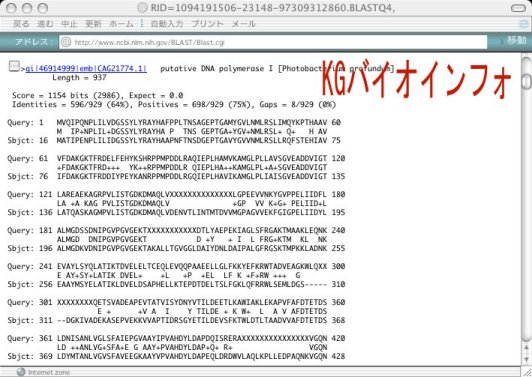
更に下へ進むと個々の配列とのアライメントが表示される。上段が問い合わせ配列 (Query) で、下段がデータベース中に見つかった配列 (Subject) であり、その間に一致したアミノ酸残基が表示される。性質の似た配列は+で表示されている。
blastpによる検索で、類似配列が見つかってもそのすべてが機能未知である場合や、得られる類似配列の数が極端に少ない場合などに試してみると良いものにPSI-BLASTがある。PSI-BLASTでは検索を繰り返し行う事によってblastpでは類似性が低く検出できなかった配列を検出することができる。PSIとはPosition-Specific Iterated (部位特異的に繰り返された) の略であり、一度blastによる検索を行った後、保存性が高く重要であると考えられるアミノ酸残基を抽出し、その配列を使用して再度検索をかけることによって類似性の低い配列を検出することを可能にしている。
操作法は、まずBLASTのホームページから"PHI- and PSI-BLAST"に入り、その後は基本的に前出のblastpと同じである。
結果表示のところまでくると、類似配列一覧のところで期待値 (E Value) が0.005より低いものに"NEW"のマークが付く。
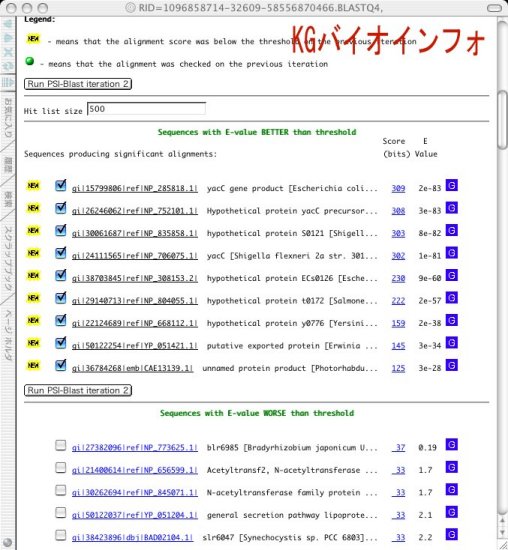
これらの"NEW"のマークが付いた配列を用いた二回目の検索を行うには"Run PSI-Blast iteration 2"のボタンをクリックする。この例では二つの"NEW"マークの付いた候補が新たにあがってきた。
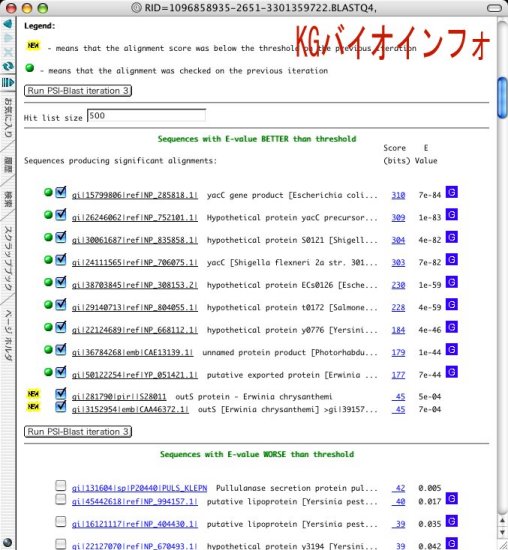
以下、これを繰り返す事により更に類似性の低い配列を検出することが可能である。ただし、"NEW"のマークが現れなくなると、それ以上検索を繰り返しても新たな配列を得ることはできない。
blastxと同様にblastpやPSI-blastのプログラムもDDBJのホームページから実行することができる (http://www.ddbj.nig.ac.jp/Welcome-j.html)。操作法などは基本的に同じであるが、blastpではドメイン構造の解析がなされない点や、PSI-blastでは前もって繰り返し回数を指定する点などが異なる。Helpのページが日本語で書かれているのでそちらも参照されたい。
機能の類似しているタンパク質間には三次構造上の類似性がみられるのが一般的である。この場合、共通しているアミノ酸配列が見いだされることが多く、モチーフ配列と呼ばれている。モチーフ配列は新しく見いだされたタンパク質の機能を推測する上で、有力な手がかりとなる。マルチプルアライメントはアミノ酸配列からこのようなモチーフ配列を抽出するのに欠くことができない。また、配列の類似しているタンパク質は共通の祖先から進化したと考えられ、これらの進化的な距離や、遺伝子の再編成など進化の過程もマルチプルアライメントから推測することができる。Clustal W はマルチプルアライメントと系統樹を作成するためのプログラムである。このプログラムでは、まず入力した配列のすべての組み合わせのペアについてアライメントを作成し、ホモロジースコアを計算する。次に、各スコアをもとに近隣結合法を用いてガイドツリー (系統樹) を作成し、各枝の長さが求められる。そして、最も近縁の配列から逐次アライメントが行われ、マルチプルアライメントが作成される。
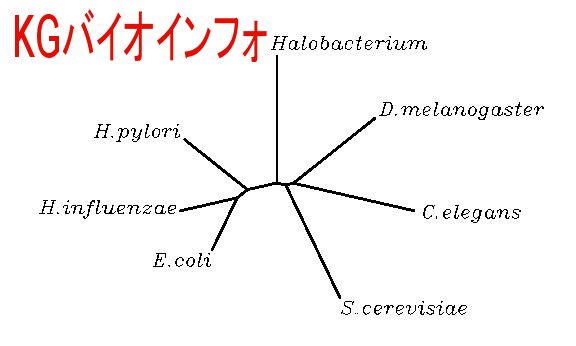
以下に示す例はアミノ酸配列を用いたマルチプルアライメントと系統樹の作成例であるが、同様にして塩基配列を用いたマルチプルアライメントや系統樹の作成も可能である。
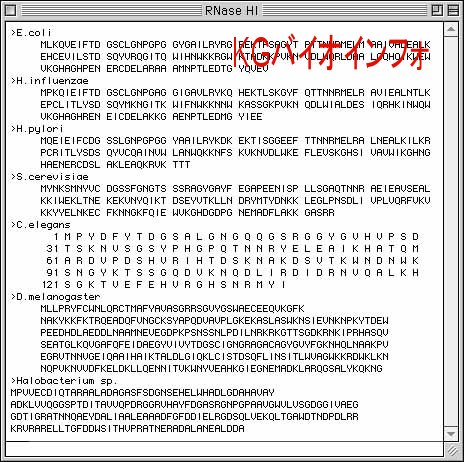
マルチプルアライメントあるいは系統樹を作成したいアミノ酸配列をテキストファイルで以下のように加工する。
"分子生物学研究用ツール集" の "配列解析" の項目のところの "clastalW" をクリック (つながらない場合は、DDBJ-CLUSTALW、EMBL-CLUSTALW"にアクセス)。
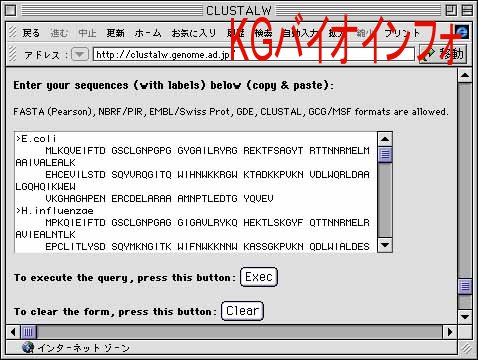
手順1で作成した内容をコピーして、配列入力用ボックスの中にペーストする。"Exec"ボタンをクリックするとマルチプルアライメントが表示される。すべての配列においてアミノ酸残基が保存されている部位は "*"、類似性の高いアミノ酸残基への置換が生じている部位は ":" 、類似性のやや低いアミノ酸残基への置換が生じている部位は "." で示されている。保存性の高い部分は活性に必要であるなど、重要な機能を担っている場合が多い。実際、例では完全に保存されている酸性アミノ酸残基が見いだされるが、これらは触媒残基である。

画面の一番下の [Unrooted N-J Tree] をクリックすると無根系統樹が表示される。"N-J" は "Neibour Joining" (近隣結合法) の略である (DDBJ-CLUSTALW、EMBL-CLUSTALWはこの機能がないので、Phylodendronを用いて系統樹を作成) 。
上述したように、モチーフとはタンパク質中で局所的に非常に良く保存されているアミノ酸配列のことであり、多くは機能と対応づけられている。一方、タンパク質はその立体構造がいくつかの構造単位に分かれていることがあり、これらの構造単位をドメインとよび、アミノ酸配列上も類似性を示すものが多い。インターネット上にはこれらモチーフやドメインに関する様々なデータベースが数多く存在している。NCBIのblastp検索やPSI-BLAST検索では類似性検索と同時にドメイン検索もできると上述したが、その際にはPfam、Smart、COGという3つのデータベースに対して検索した結果が得られる。それぞれのデータベースは相互リンクしているものも多く、たとえばPfamの結果はProsite、COG、PDBなどのデータベースの対応するファミリーにもリンクしている。そのため、基本的にはblastと同時に行うドメイン検索の結果からリンクをたどっていく事でモチーフやドメインに関する情報を十分に得る事ができる。したがって、ここではモチーフ・ドメインデータベースとそのオリジナルサイトを示す程度にとどめておく。
SOSUIは対象とする蛋白質のアミノ酸配列から、1.膜蛋白質であるかどうかの判別、2.膜貫通領域の予測、を行うプログラムである。SOSUIの最大の特徴は、配列の相同性などを利用せずに物理化学的手法を用いて99%以上という高精度で膜蛋白質を判別できることである (膜貫通領域精度は80〜95%) 。以下にその具体的な使用法を説明する。
SOSUIホームページ (http://sosui.proteome.bio.tuat.ac.jp/sosuiframe0.html)
→"SOSUI"をクリック。
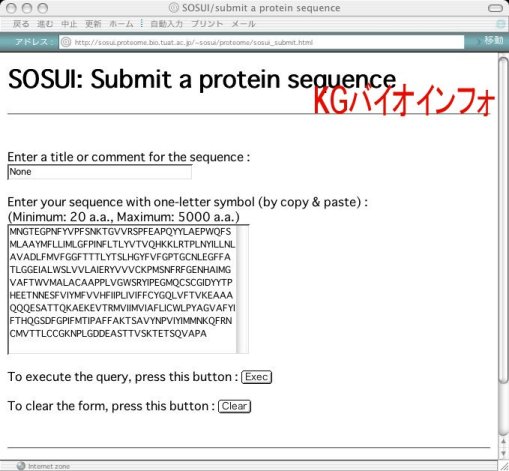
アミノ酸配列をペーストし、Execボタンをクリックして開始。
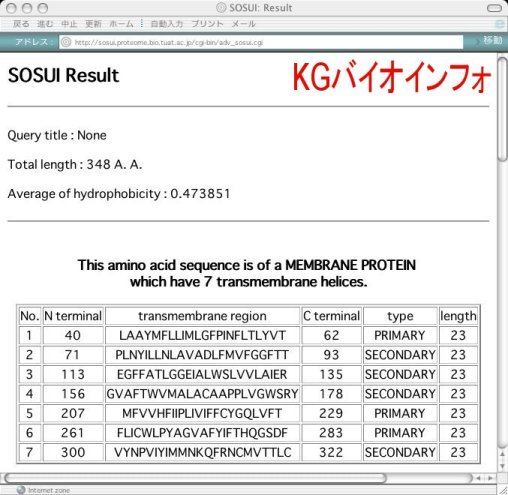
入力アミノ酸残基数と全体の平均疎水性値がヘッダ情報として出力され、続いて、膜蛋白質判別結果と膜貫通領域ヘリックス数が表示される。その下に膜貫通ヘリックスの領域とそのアミノ酸配列、ヘリックスタイプ (PRIMARY, 疎水性アミノ酸残基のみからなる; SECONDARY, 極性アミノ酸残基を含む) が表にまとめて表示される。
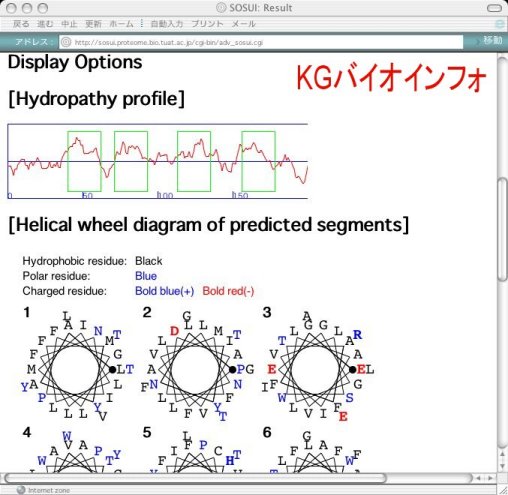
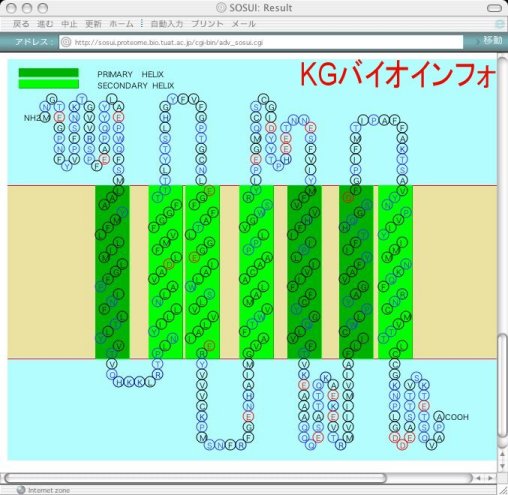
ウインドウ下部へ進むとKyte & Doolittle指標に基づく疎水性プロファイルとヘリックス車輪図が表示され、更に下へ進むとペプチド鎖の膜貫通領域が図で表示されている。