ゲノムの塩基配列が決定されただけでは、その機能についての情報は得られない。そこでまずタンパク質がどこにコードされているかを探索する必要がある。ORFは開始コドンから終止コドンまでの間がある程度の長さを持ち、タンパク質がコードされている可能性のある読み枠である。ここでは、ORF Finderを用いてORFの検索を行う。
4501 taaaaacaag ccacgaattc gccaggcggt tggagccacc cggcaatgtc gtaaaccaca
4561 ggcttaaact tcaacttggt agcctgtatc ttccagtgtg ggattcatcg ccgcggcacg
4621 agccagttca tcacagcgtt cgttttccgg gtgtccggca tggcctttaa cccattccca
4681 tttgatttga tgctgcccca atgcagcatc aagacgttgc cagagatcga cattttttac
4741 tggttttttg tctgcggttt tccagccacg ttttttccag ttatggatcc actgggtgat
4801 accctggcgg acatactggc tgtcggtact caaaatgact tcgcaatgtt cttttaacgc
4861 ctccagcgcg acaatagcgg ccatcaactc catacggttg ttggtggtgc gggtgtagcc
4921 agcgctaaag gttttctcgc gtccgcgata gcgtaaaata gcgccgtaac ccccaggtcc
4981 tggattgccc agacacgaac catcggtgaa aatttctacc tgtttaagca tctctggtag
5041 acttcctgta attgaatcga actgtaaaac gacaagtctg acataaatga ccgctatgag
"分子生物学研究用ツール集" の "配列解析" の項目のところの "ORF Finder" をクリック。

配列入力欄に目的塩基配列をペーストし、 "OrfFind" をクリックする。 ("Enter GI" or "ACCESSION" の欄にAccession numberを入力しても良い)
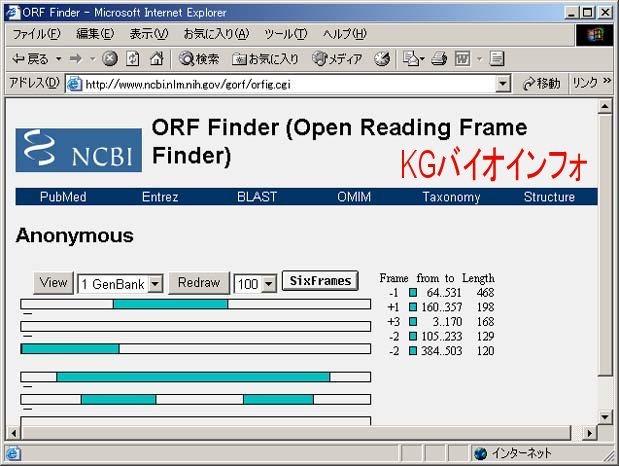
相補鎖も含めて6種類の読み枠について、見いだされたORF領域が水色で表示される。この画面では100残基以上のORFが長い順に表示されている。それぞれの領域をクリックすると、ORFとコードされたアミノ酸配列が表示される。例では、一番長い+1フレーム (コドンの読み枠) のORFに実際のタンパク質がコードされている。メチオニン残基が水色で表示され、メチオニン残基を複数含む場合は、最も上流の残基から始まるORFが表示されるが、実際の開始残基は最も上流であるとは限らない。例では最初のメチオニン残基が実際の開始残基である。開始コドンが "ATG" ではなく "GTG"、"TTG"、"CTG" である場合、"Alternative Initiation Codons" をクリックすると、これらのコドンで始まるORFが表示される。
"分子生物学研究用ツール集" の "制限酵素マップ" の項目のところの "WebCutter" をクリック。
"Paste the DNA sequence into the box below" のところに目的配列をペーストする。
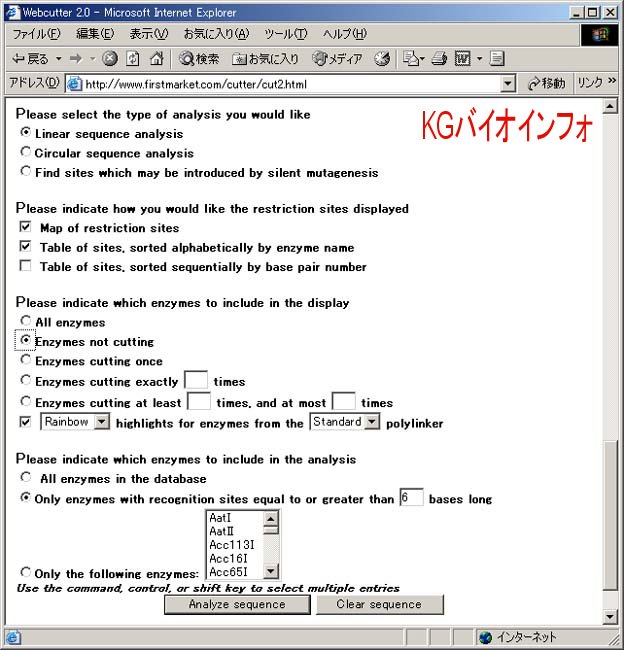
条件を設定する。上図の "Please indicate which enzymes to include in the display" のところで認識配列の数を指定する。たとえば "Enzymes cutting once" を選ぶと、配列中1カ所のみ存在する認識配列が検索できる。"Please indicate which enzymes to include in the analysis" のところで制限酵素の種類を指定する。たとえば "Only enzymes with recognition sites equal to 6 or greater than bases long" を選ぶと、認識配列が6塩基以上の酵素について検索できる。
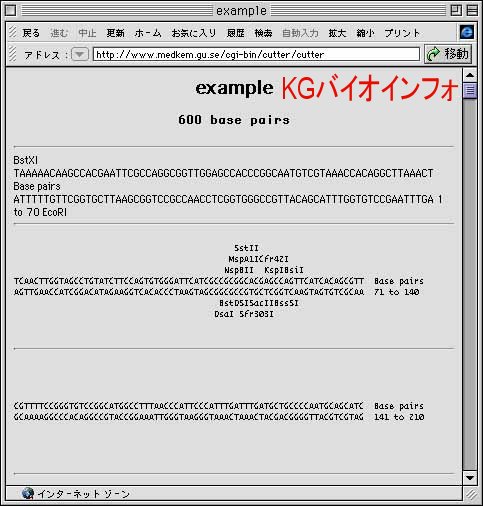
"Analyze sequence" ボタンをクリックすると、塩基配列とともに、酵素の切断部位が表示される。その下に切断部位の一覧表も表示される。
遺伝子配列を手に入れ、ORFの同定を行った後にその遺伝子がコードしているタンパク質の機能を推定するために通常まず行われるのが、ホモロジー検索 (類似配列検索、相同性検索) である。ホモロジー検索によく用いられるプログラムにblastがある。blastnは手元にある塩基配列と類似性を示す配列を塩基配列データーベースに対して検索するものであり、一方、blastxは塩基配列を6つの読み枠すべて (順方向3フレーム、逆方向3フレーム) をアミノ酸配列に変換した後、アミノ酸配列データーベースに対して検索を行うものである。ここでは例題塩基配列を用いてblastxで類似配列の検索を行った例を示す。
"分子生物学研究用ツール集" の "ホモロジー検索" の項目のところの "BLAST" をクリック (つながらない場合は、"NCBI-BLAST (http://www.ncbi.nlm.nih.gov/BLAST/)", NCC-BLAST (http://bioinfo.ncc.go.jp/BLAST/blast_pro.html)"にアクセス)。
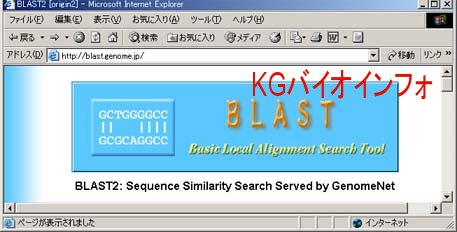
program は "BLASTX" 、database は "nr-aa" を選択。
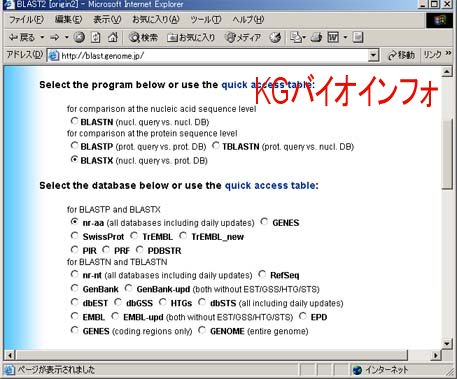
表示する sequence、alignment の数を 50 に設定。課題配列をコピーして、配列入力用ボックスの中にペーストする。"Exec"ボタンをクリック。
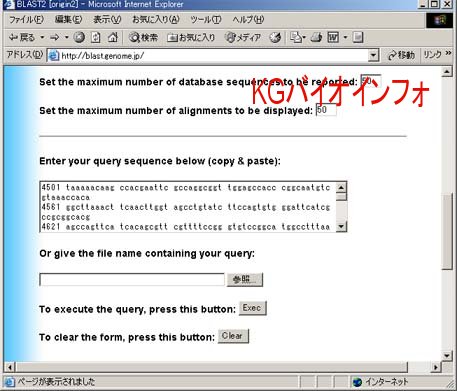
ホモロジーのスコアの高い配列から順に表示される。
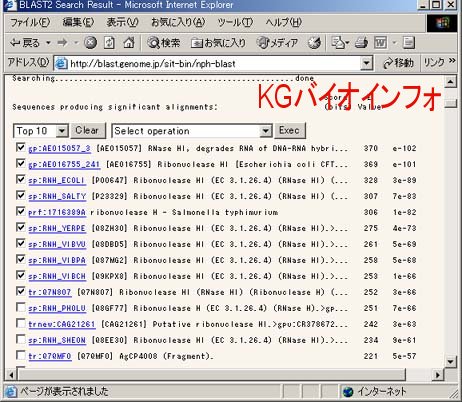
配列一覧に引き続き、スコアの高い物から順番に詳細な情報が表示される。
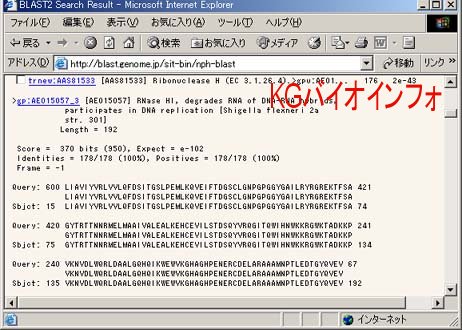
これらの情報に続き、alignment が表示される。上段が問い合わせ配列 (Query) で、下段がデータベース中に見つかった配列 (Subject) であり、その間に一致したアミノ酸残基が表示される。性質の似た残基は+で表示されている。[アクセションNo.] のリンクへつなぐと、関連する論文や全アミノ酸配列など、さらに詳細な情報を得ることができる。